Structural Multiplex cfDNA Reference Standard, 350 ng
O Padrão de Referência de cfDNA Multiplex Estrutural fornece material de controle de qualidade biologicamente relevante, que pode ser usado para avaliar o desempenho de ensaios NGS ctDNA que detectam variantes estruturais complexas.
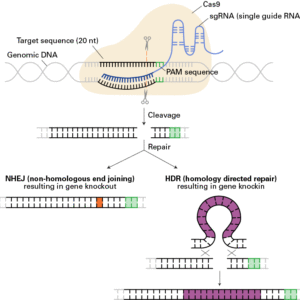
Variantes estruturais, incluindo translocações de genes, fusões, variantes de número de cópias (CNV) e grandes INDELs, são comumente encontradas em tumores e podem servir como biomarcadores clínicos para identificar o câncer, monitorar a eficácia do tratamento do câncer e a progressão da doença. CNVs como a amplificação de MET em combinação com mutações em EGFR (T790M) podem levar à resistência ao gefitinibe no câncer de pulmão ( 1,2 ) . Ensaios baseados em PCR ( 3 , 4 ) e NGS ( 4) que melhor detectam variantes estruturais estão constantemente surgindo. No entanto, a detecção de variantes estruturais apresenta vários desafios e os pipelines de bioinformática estão evoluindo continuamente para melhorar a confiança na detecção de variantes estruturais. O padrão de referência cfDNA Multiplex estrutural cobre uma ampla gama de mutações em um contexto genômico definido. Este padrão consiste em DNA derivado de linhas de células humanas que é fragmentado em um tamanho médio de 160 pb para se assemelhar ao cfDNA extraído do plasma humano. Este produto foi projetado para desafiar seu fluxo de trabalho molecular e bioinformático, fornecendo variantes/amplificações de número de cópias validadas, translocações e grandes inserções/exclusões. Além disso, você pode examinar o contexto genômico de variantes em regiões de conteúdo GC específico (alto x baixo). O Padrão de Referência de cfDNA Multiplex Estrutural inclui 9 variantes validadas por ddPCR, com a maioria delas centradas em 5% de frequência alélica. Os recursos de destaque do multiplex estrutural incluem variantes de fusão RET e ROS1, amplificações focais MYC-N e MET e uma variante BRCA2. O Multiplex estrutural também está disponível em gDNA de alto peso molecular (HD753 ) e FFPE ( HD789 ).
Com este produto você é capaz de:
- Avalie o efeito do contexto genômico na detecção de variantes em amostras de ctDNA
- Analise a robustez do seu pipeline de bioinformática
- Otimize e valide novos fluxos de trabalho de ctDNA para detecção de variantes estruturais
- Compare o desempenho de padrões de referência fragmentados com de alto peso molecular
Dados técnicos
Formato: cfDNA
Genes abrangidos: AKT1, BRCA2, EGFR, FBXW7, FLT3, GNA11, KRAS, MET, MYC-N, NOTCh2, PIK3CA, RET, ROS1
Cósmicos: 1.500+
Frequências alélicas: 4,8-5,6% e amplificação de 4,5 e 9,5 x para o CNV
Tampão: Tris-EDTA (10mM Tris-HCl, 1mM EDTA), pH 8,0
Produtos relacionados
-
Leia mais
O padrão de referência BRCA Somatic Multiplex 1 no formato FFPE é um material de controle de qualidade altamente caracterizado e biologicamente relevante usado para…
-
R$9.128,76Adicionar ao carrinho
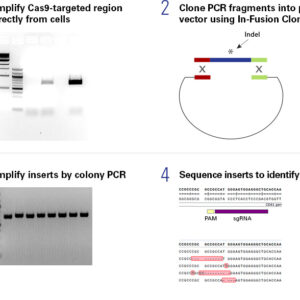
O Guide-it Indel Identification Kit é utilizado para caracterização de inserções e deleções (indels) geradas por ferramentas de edição de genes, como CRISPR/Cas9. Este kit contém…
-
R$8.390,79Adicionar ao carrinho
Controles para verificar quebras de fita dupla de DNA e eficiências de edição de genes Validado Edit-R Todos-em-um controles positivos lentivirais sgRNA e iniciadores de…
-
R$15.220,59Adicionar ao carrinho
Guide-it Cas9 recombinante (10 µg/µl) é uma nuclease de Streptococcus pyogenes Cas9 recombinante de tipo selvagem expressa com um sinal de localização nuclear C-terminal (NLS), fornecido em…