RNase A (Lyophilized Powder), 100mg
R$319,44
RNase A
N9046 (100MG)
Atividade específica: >2.500 u/mg proteínas (>units/mg proteínas)
Formato: contém 100mg de RNase A liofilizada.
Armazenar em temperatura ambiente por até um ano. Para um período maior armazenar em 4°C.
Descrição
RNase A, livre de DNase e proteases é uma endoribonuclase que degraga especificamente fitas únicas de RNA, clivando as ligações de fosfodiéster entre 5’ ribose do nucleotídeo e o grupo fosfato anexado a 3’ ribose de um nucleotídeo de pirimidina adjacente. O 2′, 3′-fosfato cíclico resultante é hidrolisado ao 3′ nucleosídeo fosfato correspondente.
Aplicações
Preparação de DNA genomico e plasmídeos
Remoção do RNA a partir de amostras de proteína recombinante
Ensaios de proteção à ribonuclease
Mapeamento de mutações de uma única base de DNA ou RNA
Controle de Qualidade
A ausência de endodesoxirribonucleases, exodeoxiribonucleases e proteases foi confirmada por testes de qualidade adequados. Funcionalmente testadas para a digestão de RNA em um procedimento de purificação de DNA plasmidial.
Fonte
Pâncreas bovino.
Peso Molecular
13,7 kDa monómero.
Definição de Atividade por Unidade
Uma unidade da enzima provoca um aumento na absorbância de 1,0 a 260nm quando RNA de levedura é hidrolisado a 37°C e pH 5.0
Cinquenta unidades equivalem a aproximadamente equivalente a 1 unidade de Kunitz
Inibição e Inativação
Inibidores: O inibidor mais potente é uma proteína de ~50 kDa a partir de citosol de células de mamíferos, por exemplo, RiboLock™ RNase inibidor.
Outros inibidores: uridina 2′, 3′ vanadato cíclico, 5′-difosfoadenosina 3′-fosfato e 5′-difosfoadenosina 2′-fosfato (2), SDS, dietil pirocarbonato, guanidínio tiocianato 4M mais 0,1 M de 2-mercaptoetanol e íons de metais pesados. Inativado por extração com fenol / clorofórmio.
Inativado por extração com fenol/clorofórmio.
Inativado por aquecimento a 95°C durante 10 minutos.
Disponível por encomenda
| Nota A concentração ótima para RNase A é de 1-100 ug/ml, dependendo da aplicação. |
| A enzima é ativa sob uma grande variedade de condições de reação. Em baixas concentrações de sal (NaCl 0 a 100 mM), a RNase A cliva o RNA de cadeia simples e de cadeia dupla, bem como a cadeia de RNA em híbridos de RNA-DNA. No entanto, em concentrações de NaCl de 0.3M ou superior, a RNase A cliva especificamente o RNA de cadeia simples. |
Produtos relacionados
-
R$266,20Adicionar ao carrinho
Descrição
Proteinase K pronta pra usar é uma protease endolítica que cliva ligações peptídicas nos lados carboxílicos de aminoácidos alifáticos, aromáticos ou hidrofóbicos.
A Proteinase K é classificada como uma serina protease. O menor peptídeo a ser hidrolisado por essa enzima é um tetrapeptídeo.Aplicações
- Isolamento de DNA genômico de células e tecidos cultivados;
- Remoção de DNases e RNases ao isolar DNA e RNA de tecidos celulares;
- Melhorar a eficiência de clonagem de produtos de PCR.
Concentração
20 mg /mlBuffer de Armazenamento
A enzima é fornecida em: Tris-HCl 50 mM (pH 7,5), contendo cloreto de cálcio 5 mM e glicerol a 50% (v/v).Inibição e Inativação
Inibidores: a proteinase K não é inativada por quelantes de metal, por reagentes reativos com tiol ou por inibidores específicos de tripsina e quimiotripsina. O fluoreto de fenilmetilsulfonil e o diisopropil fosforofluoridato inibem completamente a enzima.
Inativado por aquecimento a 95 °C por 10 minutos.Observação
- Atividade ótima em 50-55 ° C.
- A desnaturação rápida da enzima ocorre em temperaturas acima de 65 ° C.
- A concentração de trabalho recomendada para Proteinase K é de 0,05-1 mg /ml. A atividade da enzima é estimulada por SDS 0,2-1% ou por uréia 1-4 M.
- O Ca2 + protege a Proteinase K contra a autólise, aumenta a estabilidade térmica e tem uma função reguladora para o local de ligação do substrato da Proteinase K.
- Estável em uma ampla faixa de pH: 4,0-12,5, pH ideal 7,5-8,0
-
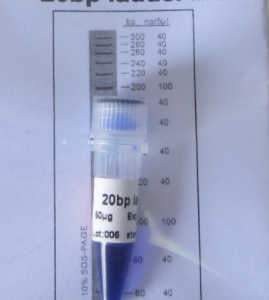
R$702,77O preço original era: R$702,77.R$450,00O preço atual é: R$450,00.Adicionar ao carrinho20bp DNA ladder
M1021 (50UG)Concentração: 128 ng/ul
Armazenar a -20°CVolume estimado: 500ul
Descrição
20bp DNA ladder é ideal para determinar o tamanho de fitas duplas de DNA de 60 a 300 pares de base. O marcador consiste em 13 fragmentos de dupla fita. Os fragmentos de tamanhos 100 e 200bp estão presentes com maior intensidade para permitir fácil identificação. Todos os fragmentos são precisamente quantificados e misturados durante a fabricação. Para carregamento de 5ul todos os fragmentos têm 40ng, com exceção dos fragmentos 100 e 200bp que têm 100ng. Este marcador é pré-misturado com tampão de carregamento azul e está pronto para uso.Carregamento recomendado
2-5 ul por poçoConcentração
Bandas em evidência 100 ng/5ul
Outras bandas 40 ng/5ulCondições recomendadas para Eletroforese
Gel de poliacrilamida 10%, 20 cm, 1X TBE, 8 V/cm, 3h.Conteúdo (bp)
60, 80, 100, 120, 140, 160, 180, 200, 220, 240, 260, 280, 300. -
R$425,92Adicionar ao carrinho
M7012 – DS View Nucleic Acid Stain, 20.000X
Embalagem: 500 ul
Concentração: 20.000xO DSView é uma alternativa ao tradicional corante de brometo de etídio (EB) para detecção de ácido nucléico em géis de agarose. Emite fluorescência verde quando ligado ao DNA ou RNA. O corante DSView tem dois máximos de excitação de fluorescência: em 267 nm e outro em 294 nm. Além disso, também tem uma excitação visível a 491nm.
Armazenamento: guarde a 4ºC por até 2 anos.
Protocolo
1. Preparar 100 ml de solução de gel de agarose (concentração de 0,8 a 2%) e misturar completamente. Coloque o frasco no microondas, aqueça até que a solução esteja completamente limpa e não haja partículas flutuantes pequenas visíveis (cerca de 2 ~ 3 minutos).
2. Adicione 2-5μl de DSViewTM à solução de gel. Agite o frasco suavemente para misturar a solução e evite formar bolhas.
3. Enquanto a solução de gel esfria, despeje-a na bandeja de gel até que os dentes do pente estejam imersos cerca de 1/4 ~ 1/2 na solução de gel.
4. Permitir o gel de agarose esfriar até solidificado. Coloque amostras no gel e corra a eletroforese.
5.Detectar as bandas sob iluminação UV.
-
R$574,99Adicionar ao carrinho
Descrição
Marcador para determinar o tamanho do DNA de fita dupla de 25 a 700 pares de bases.
O ladder consiste em 10 fragmentos lineares de fita dupla. Os fragmentos de 100pb e 300pb estão presentes em intensidade aumentada para permitir fácil identificação.
Todos os fragmentos são quantificados com precisão e misturados durante a produção.Para carregamento de 5ul, todos os fragmentos, exceto 100bp e 200bp, são de 40 ng. O fragmento de 100 pb e 200 pb é de 100 ng.
O marcador é pré-misturado com corante e está pronto para uso.
Carregamento recomendado
2-5 UL
Concentração
Banda em evidência 100 ng/5ul
Outras bandas 40 ng/5ulConteúdo (bp)
25, 50, 75, 100, 150, 200, 300, 400, 500, 700