DS View Nucleic Acid Stain, 20.000X, 1 ml
R$1.053,90
M7011 – DS View Nucleic Acid Stain, 20.000X
Embalagem: 1 ml
Concentração: 20.000x
O DSView é uma alternativa ao tradicional corante de brometo de etídio (EB) para detecção de ácido nucléico em géis de agarose. Emite fluorescência verde quando ligado ao DNA ou RNA. O corante DSView tem dois máximos de excitação de fluorescência: em 267 nm e outro em 294 nm. Além disso, também tem uma excitação visível a 491nm.
Armazenamento: guarde a 4ºC por até 2 anos.
Protocolo
1. Preparar 100 ml de solução de gel de agarose (concentração de 0,8 a 2%) e misturar completamente.
Coloque o frasco no microondas, aqueça até que a solução esteja completamente limpa e não haja partículas flutuantes pequenas visíveis (cerca de 2 ~ 3 minutos).
2. Adicione 2-5ul de DSViewTM à solução de gel. Agite o frasco suavemente para misturar a solução e evite formar bolhas.
3. Enquanto a solução de gel esfria, despeje-a na bandeja de gel até que os dentes do pente estejam imersos cerca de 1/4 ~ 1/2 na solução de gel.
4. Permitir o gel de agarose esfriar até solidificado. Coloque amostras no gel e corra a eletroforese.
5. Detectar as bandas sob iluminação UV.
Disponível por encomenda
| Nota |
| 1. A espessura do gel deve ser inferior a 0.5 cm, uma vez que os géis espessos podem diminuir a sensibilidade. |
| 2. O derretimento repetido dos géis contendo o DSViewTM pode resultar em baixa sensibilidade. |
| 3. DSViewTM permite a visualização de DNA (10ng) no gel de agarose sob luz visível. Isso elimina a necessidade de exposição à luz UV, que pode cortar e danificar o DNA. Os fragmentos de DNA intactos purificados a partir de gel de agarose podem aumentar a eficiência de manipulações subsequentes da biologia molecular, tais como clonagem, transformação e transcrição. |
| 4. O DSViewTM pode irritar a pele e os olhos. Por favor, use luvas durante o manuseio. |
| 5. Detecção de sensibilidade do DSViewTM sob transmissão UV (comprimento de onda 300nm). |
Produtos relacionados
-
R$2.213,19Adicionar ao carrinho
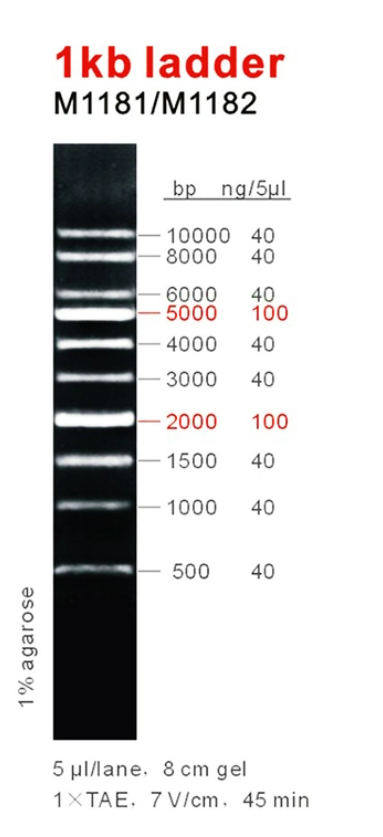
1kb DNA ladder
M1182 (5x50ug)
Concentração: 104ng/ul
Armazenar a -20°C
Volume estimado: 5x500ul
Descrição
1kb DNA ladder é ideal para determinar o tamanho de fitas duplas de DNA de 500 a 10.000 pares de base. O marcador consiste em 10 fragmentos de dupla fita. Os fragmentos de tamanho 2.000 e 5.000bp estão presentes com maior intensidade para permitir fácil identificação. Todos os fragmentos são precisamente quantificados e misturados durante a fabricação. Para carregamento de 5ul todos os fragmentos têm 40ng, com exceção dos fragmentos 2.000 e 5.000bp que têm 100ng. Este marcador é pré-misturado com tampão de carregamento azul e está pronto para uso.
Carregamento recomendado
2-5 ul por poço
Concentração
Bandas em evidência 100 ng/5ul
Outras bandas 40 ng/5ul
Condições recomendadas para Eletroforese
Gel de agarose 1%, 8cm, 1X TAE, 7 V/cm, 45 min.
Conteúdo (bp)
500, 1.000, 1.500, 2.000, 3.000, 4.000, 5.000, 6.000, 7.000, 8.000 e 10.000 -
R$1.053,90Adicionar ao carrinho
Solução RNase A
Grau BR, somente para uso em pesquisa.
Nº N9042 Concentração: 100 mg/ml Tamanho: 1 ml
Atividade Específica: ≥3000 U/mg de proteína (≥60 unidades Kunitz/mg de proteína).
Componentes
Componente N9041 ( 10mg/ml) N9042 (1 00mg/ml) Solução RNase A 1 ml 1 ml Descrição
A RNase A é uma endorribonuclease que degrada especificamente o RNA de fita simples nos resíduos C e U. Ele cliva a ligação fosfodiéster entre a ribose 5′ de um nucleotídeo e o grupo fosfato ligado à ribose 3′ de um nucleotídeo pirimidina adjacente. O fosfato 2′,3′-cíclico resultante é hidrolisado no fosfato 3′-nucleosídeo correspondente.Formulários
-Preparação de plasmídeo e DNA genômico
-Remoção de RNA de preparações de proteínas recombinantes.
–Ensaios de proteção de ribonuclease
–Mapeamento de mutações de base única em DNA ou RNAArmazenar _
-20ºC recomendado.
Monômero de peso molecular
13,7 kDa. -
R$526,95Adicionar ao carrinho
1kb DNA ladder plus
M1191 (50ug)Concentração: 144ng/ul
Armazenar a -20°C
Volume estimado: 500ulDescrição
1kb DNA ladder plus é ideal para determinar o tamanho de fitas duplas de DNA de 100 a 10.000 pares de base. O marcador consiste em 15 fragmentos de dupla fita. Os fragmentos de tamanho 500 e 3.000bp estão presentes com maior intensidade para permitir fácil identificação. Todos os fragmentos são precisamente quantificados e misturados durante a fabricação. Para carregamento de 5ul todos os fragmentos têm 40ng, com exceção dos fragmentos 500 e 3.000bp que têm 100ng. Este marcador é pré-misturado com tampão de carregamento azul e está pronto para uso.Carregamento recomendado
2-5 ul por poçoConcentração
Bandas em evidência 100 ng/5ul
Outras bandas 40 ng/5ulCondições recomendadas para Eletroforese
Gel de agarose 1%, 8cm, 1X TAE, 7 V/cm, 45 min.Conteúdo (bp)
100, 200, 300, 400, 500, 700, 1.000, 1.500, 2.000, 3.000, 4.000, 5.000, 6.000, 8.000 e 10.000 -
R$927,43Adicionar ao carrinho
Descrição
O DNA Ladder de 10 pb é ideal para determinar o tamanho do DNA de fita dupla de 80 a 300 pares de bases. A marcador consiste em 18 fragmentos lineares de fita dupla.
Os fragmentos de 100 pb e 200 pb estão presentes em intensidade aumentada para permitir fácil identificação. Todos os fragmentos são quantificados com precisão e misturados durante a produção. Para carregamento de 5 ul, todos os fragmentos, exceto 100 pb e 200 pb, são de 40 ng.
Os fragmentos de 100 pb e 200 pb são 100 ng. Este marcador é pré-misturado com corante e está pronto para uso.Carregamento recomendado
2-5 ulConcentração
Bandas em evidência 100 ng / 5 ul
Outras bandas 40 ng / 5 ulCondição de eletroforese recomendada
1 ul / por poço, 20 cm, gel de poliacrilamida a 10%, 1 × TBE, 8 V / cm, 3h.Conteúdo (bp)
80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 220, 240, 260, 280, 300.Concentração
168 ng / ul