Gel Loading Dye, Three-colour (6X), 5x1ml
R$175,65
6 × corante de carregamento de gel , três cores
M9061 Tamanho: 1 ml × 5
Descrição
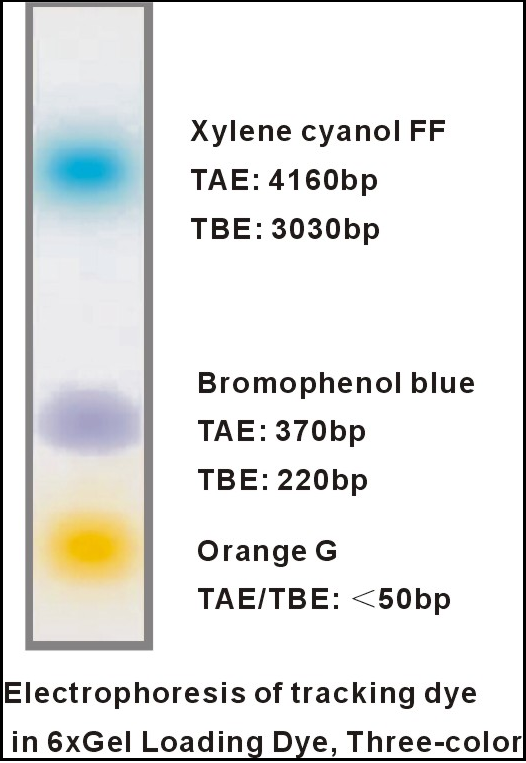
6× Gel Charging Dye, Three-Color é um tampão de carga pré-misturado com três corantes de rastreamento para eletroforese em gel de agarose e poliacrilamida não desnaturante. O EDTA é incluído para quelar o magnésio (até 10 mM) em reações enzimáticas, interrompendo assim a reação. Azul de bromofenol, xileno cianol e laranja G são os corantes de rastreamento padrão para eletroforese.
Conteúdo
Tris-HCl 10 mM (pH 7,6), 0,03% de xileno cianol FF, 0,03% de azul de bromofenol, 0,15% de laranja G, 60% de glicerol, EDTA 60 mM.
Aplicação
Preparação de escadas de DNA, marcadores e amostras para carregamento em géis de agarose ou poliacrilamida.
Características
Rastreamento de três cores da migração de DNA durante a eletroforese.
Sem mascaramento de DNA durante a exposição do gel à luz UV.
O EDTA liga-se a íons metálicos divalentes e inibe nucleases dependentes de metal.
Armazenar
Armazenar em temperatura ambiente ou a 4°C por até 12 meses. Para períodos mais longos, armazenar a -20 ̊C.
Disponível por encomenda
Declaração de garantia de qualidade
O corante de carregamento de gel, três cores (6×) é testado quanto à ausência de endonuclease, exonuclease e nenhuma atividade de RNase.
Produtos relacionados
-
R$2.459,10Adicionar ao carrinho
Descrição
O marcador 50 pb é ideal para determinar o tamanho do DNA de fita dupla de 50 a 500 pares de bases. A escada consiste em 8 fragmentos lineares de fita dupla. O fragmento de 250 pb está presente em intensidade aumentada para permitir fácil identificação. Todos os fragmentos são quantificados com precisão e misturados durante a produção.
Para carregamento de 5 ul, todos os fragmentos, exceto 250bp, são de 40 ng. O fragmento de 250 pb é de 100 ng.
O marcador é pré-misturado com corante e está pronto para uso.
Carregamento recomendado
2-5 ul
Concentração
Banda em evidência 100 ng / 5 ul
Outras bandas 40 ng / 5 ulCondição de eletroforese recomendada
8 cm, Gel de agarose a 3%, 0,5 × TBE, 5 V/cmConteúdo (bp)
50, 100, 150, 200, 250, 300, 400, 500 -
R$579,65Adicionar ao carrinho
Descrição
O marcador 200 bp é ideal para determinar o tamanho do DNA de fita dupla de 200 a 4.000 pares de bases. O ladder consiste em 12 fragmentos lineares de fita dupla.
O fragmento de 1.000 pb está presente em intensidade aumentada para permitir fácil identificação. Todos os fragmentos são quantificados com precisão e misturados durante a produção. Para carregamento de 5 ul, todos os fragmentos, exceto 1.000 bp, são de 40 ng. O fragmento de 1.000 bp é de 100 ng. Este marcador é pré-misturado com tampão de carregamento azul e está pronto para uso.Carregamento recomendado
2-5 ul / poço
Concentração
Bandas em evidência 100 ng /5ul
Outras bandas 40 ng /5ul
Condição de eletroforese recomendada
8 cm, Gel de Agarose a 1%, 1 × TAE, 7 V / cm, 40min.
Conteúdo (bp)
200, 400, 600, 800, 1.000, 1.200, 1.400, 1.600, 1.800, 2.000, 2.200, 4.000.
Concentração
108 ng /ul
Estável por 3 meses em temperatura ambiente, para armazenamento a longo prazo, armazene a -20 ℃.
-
R$312,66Adicionar ao carrinho
Descrição
dTTP (2′-desoxiadenosina 5′-trifosfato) é fornecido como uma solução aquosa 100 mM a pH 7,0.Aplicações
Para uso em PCR, PCR longo, RT-PCR, síntese de cDNA, extensão de primer, sequenciamento de DNA, marcação de DNA. -
R$158,09Adicionar ao carrinho
Conteúdo
Gel Loading Dye, Blue (6X):10 mM Tris-HCL (pH 7.6)
0.03% Bromophenol Blue
60% Glycerol
60 mM EDTADescrição
Gel Loading Dye, Blue (6X) é um tampão de carregamento pré-misturado com um corante de rastreamento para eletroforese em gel de agarose e poliacrilamida não desnaturante.
EDTA é incluído para quelar magnésio (até 10 mM) em reações enzimáticas, parando assim a reação. O azul de bromofenol é o corante de rastreamento padrão para eletroforese.
Declaração de garantia de qualidade
Gel Loading Dye, Blue (6X) é testado para contaminação de endonuclease, exonuclease e atividade RNase.Taxa de migração
O azul de bromofenol migra a aproximadamente 370 bp em um gel de agarose TAE a 1% padrão e 220 bp em um gel de agarose TBE a 1% padrão.