SMART-Seq® Total RNA Pico Input with UMIs (ZapR® Mammalian) 24 Rxns
R$29.273,13
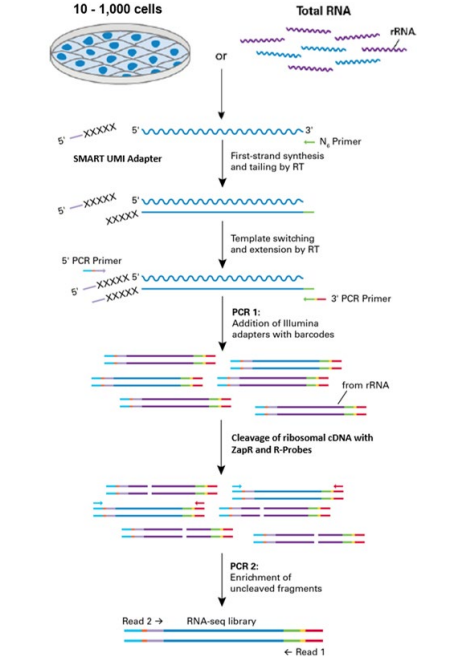
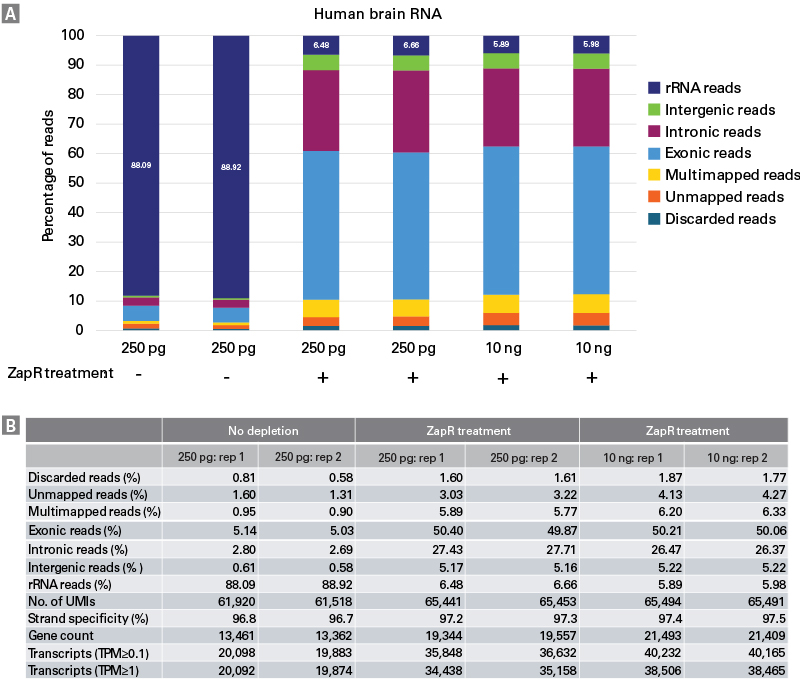
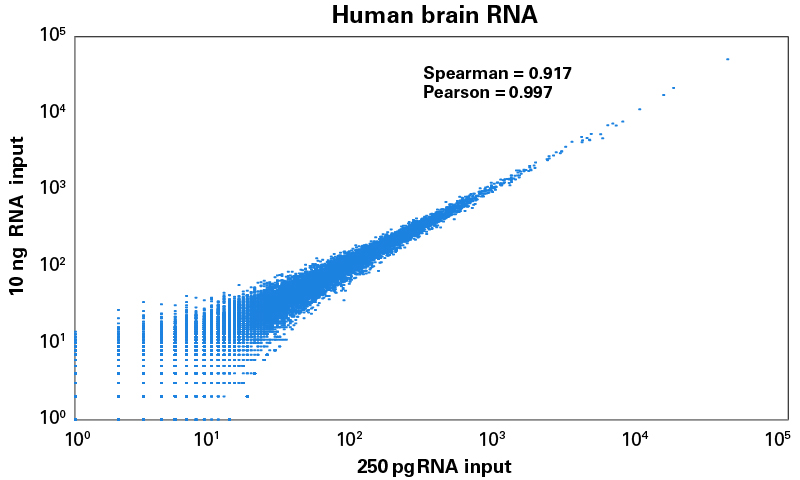
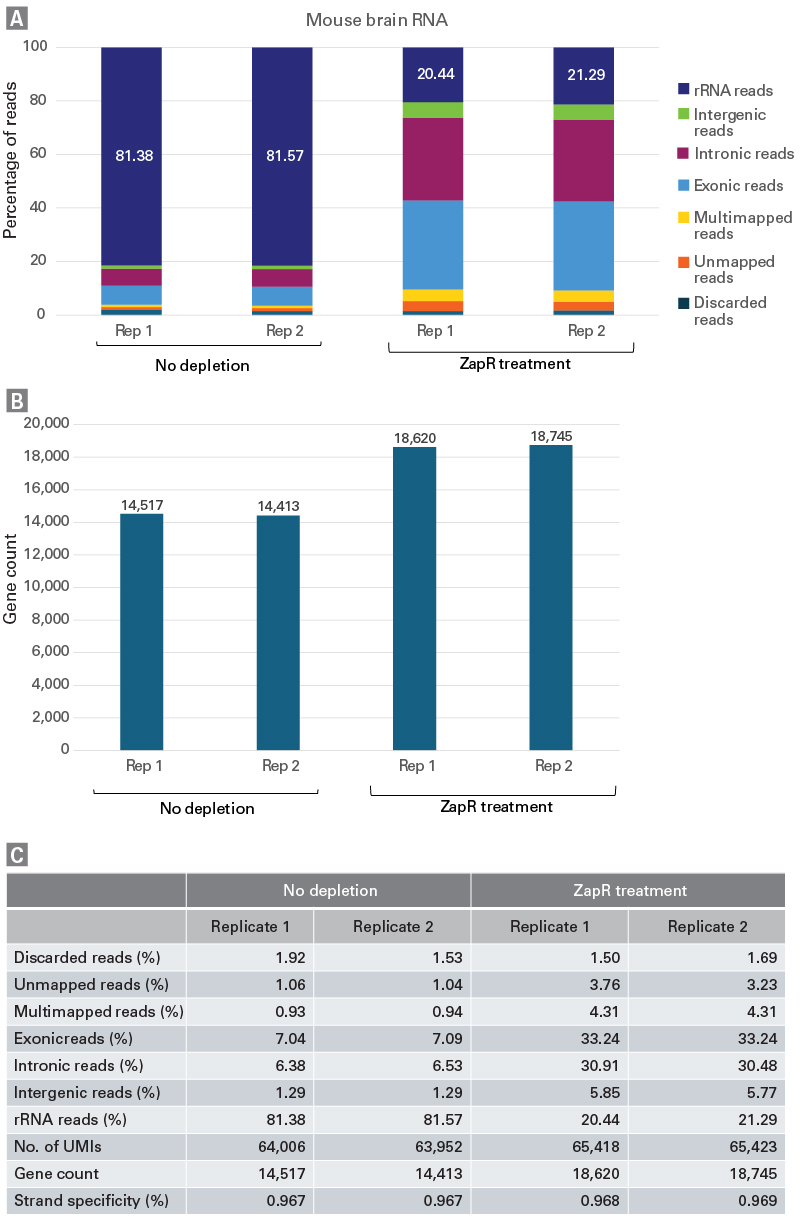
SMART-Seq Total RNA Pico Input com UMIs (ZapR Mammalian) gera bibliotecas de RNA-seq específicas de fita para sequenciamento Illumina a partir de entradas de 250 pg–10 ng de RNA total purificado ou de 10–1.000 células intactas. O design da biblioteca apresentado neste kit adiciona um identificador molecular exclusivo (UMI) de 8 nucleotídeos (nt) por meio da etapa de transcrição reversa para mitigar o potencial viés de PCR, bem como fornecer informações adicionais para quantificação de transcrição. Material suficiente é fornecido com este kit para realizar até 24 reações.
Disponível por encomenda
Produtos relacionados
-
R$15.418,26Adicionar ao carrinho
TaKaRa Ex Taq DNA Polymerase combina o desempenho comprovado da Takara Taq polimerase com a atividade de revisão de uma exonuclease 3′-5 ‘eficiente, para reações de PCR de alta sensibilidade e alta eficiência. Também pode ser usado para PCR de longo alcance (até 20 kb de modelos de DNA genômico e até 30 kb de modelos de DNA lambda). A polimerase Ex Taq tem uma fidelidade maior do que a Taq padrão com uma taxa de mutação aproximadamente 4,5 vezes menor, conforme determinado pelo método de Kunkel. A polimerase Ex Taq é fornecida com tampão 10X otimizado (com ou sem Mg 2+ ) e dNTPs.
-
R$8.309,33Adicionar ao carrinho
O meio NDiff 227 é um meio definido, isento de soro, suplementado com N2 e B-27 para diferenciação neural de células-tronco embrionárias (ES) de camundongo em monocultura aderente (Ying et al. 2003). O NDiff 227 também pode ser suplementado para permitir a cultura de células-tronco pluripotentes de longo prazo. A retirada sucessiva dos suplementos resulta na diferenciação na linhagem neural.
-
R$5.355,06Adicionar ao carrinho
O Chaperone Plasmid Set consiste em cinco plasmídeos diferentes, cada um dos quais é projetado para expressar múltiplas chaperonas moleculares que funcionam como uma “equipe de chaperone” para permitir o dobramento de proteínas. A co-expressão de uma proteína alvo com uma dessas equipes de chaperonas aumenta a recuperação de proteínas solúveis. Cada plasmídeo carrega uma origem de replicação derivada de pACYC e um gene Cm r , que permite o uso com sistemas de expressão de E. coli utilizando plasmídeos do tipo ColE1 contendo um gene de resistência à ampicilina como marcador. Os genes chaperone estão situados a jusante de um araB ou Pzt-1(tet) promotor. Portanto, a expressão de proteínas alvo e chaperonas pode ser induzida individualmente se o gene alvo for colocado sob o controle de outros promotores (por exemplo, lac ). Esses plasmídeos também contêm o regulador necessário ( araC ou tetr ) para cada promotor.
Use este conjunto para realizar um método de duas etapas para construir sistemas de co-expressão de alvo/chaperona: Na primeira etapa, prepare um hospedeiro E. coli transformado apenas com o plasmídeo chaperona. O segundo passo é preparar células competentes a partir desta nova estirpe e transformar esta estirpe com um plasmídeo que expressa a proteína alvo.
-
R$3.611,97Adicionar ao carrinho
Agarose de baixo ponto de fusão para separar fragmentos de DNA de 1 kb ou mais. Pode ser usado para recuperar fragmentos a serem usados em reações enzimáticas, como digestão de restrição, ligação e PCR.